南湖新闻网讯(通讯员 钱胜)5月27日,我校生命科学技术学院、生物医学与健康学院精准营养与代谢团队陈振夏课题组在国际学术期刊Briefings in Bioinformatics在线发表最新研究成果,该研究基于机器学习的算法开发了可自动下载和筛选高通量测序数据的软件MassiveQC,并将其应用于黑腹果蝇的研究,通过系统整合该物种所有公开RNA-seq数据,解析基因表达动态性的调控规律和演化模式。该研究为后续其他物种的表达调控分析提供了基础。

MassiveQC工作原理
高通量RNA-seq数据的爆炸式增长为各种生物学过程提供了新的方法和见解。然而由于数据异质性等问题,在整合时仍难以保证数据的质量。虽然已经开发了一些质量控制方法,但这些方法很少考虑样本间的一致性,且容易受到人为因素的影响。为此,本研究开发了基于无监督机器学习的软件MassiveQC,用于自动下载和过滤大规模高通量数据。与其他软件相比,MassiveQC使用了机器学习的方法,从样本群中提取多种重要的特征,然后基于所有样本的特征进行隔离森林的模型训练并筛选离群值。由于筛选的截止点是模型根据总样本生成,因此对用户很友好,并且适用于多物种多模式数据。
随后,本研究将MassiveQC应用于黑腹果蝇的研究,下载6万个RNA-seq样本并生成了一个包含28个组织从胚胎发育到成年的综合转录组图谱。本研究定量分析了每个基因的表达动态性,发现不同基因的表达动态性具有显著的差异。结果显示,具有低表达动态的基因很可能在演化上是古老的,并倾向于在发育早期表达,表现出低的非同源替代率,较高的突变致死率,并涉及复杂的转录和转录后调控通路,表明这些基因受到选择限制,参与核心的调控过程。
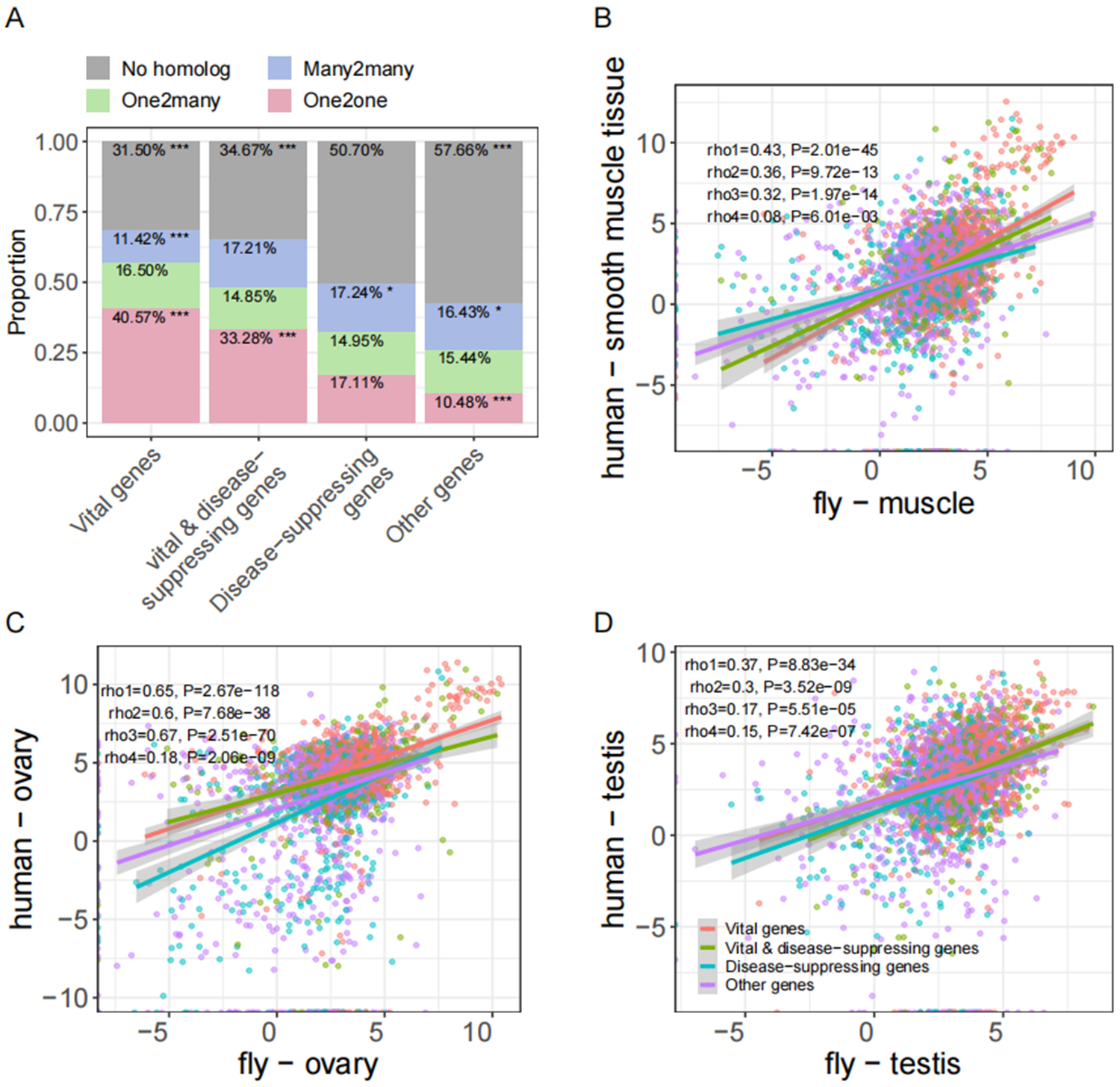
果蝇和人类在基因种类和表达量上的保守性
研究人员还分析了果蝇和人类在基因种类上的保守性,发现具有致死性和与疾病相关的基因在果蝇和人之间有更高的保守性,表达上相关性也更高,为后续果蝇在人类医学上的应用奠定了理论和数据基础。
据悉,我校生命科学技术学院、生物医学与健康学院、湖北洪山实验室博士生钱胜、石梦威、王丹阳和美国国立卫生研究院博士后Justin Fear为论文第一作者,美国国立卫生研究院Brian Oliver研究员和我校陈振夏教授为论文通讯作者。该项工作得到了国家自然科学基金、湖北省重大科技专项基金、湖北洪山实验室基金、中央高校基本科研专项资金、华中农业大学科技自主创新基金等项目的资助。
审核人陈振夏
