南湖新闻网讯(通讯员 尤佳琦 刘振平)10月16日,《自然—遗传》杂志(Nature Genetics)在线发表了我校棉花遗传改良团队的研究论文“Regulatory controls of duplicated gene expression during fiber development in allotetraploid cotton”。该研究解析了异源四倍体陆地棉两个亚基因组对纤维发育和品质形成的动态协同调控机制,阐释了亚基因组同源基因用于纤维品质改良的潜力,提出了基因组模块化的优质棉设计育种构想,创新了多倍体性状优势形成的调控理论和育种理念。
多倍化是植物演化的重要推动力,异源多倍体在自然界广泛存在。异源四倍体陆地棉是当今全球最重要的棉花栽培种,是最重要的天然纺织纤维来源,也是多倍化机制研究的典型作物。每年我国进口优质棉纤维占消耗总量约2/3,培育高品质陆地棉品种是产业的重要需求。由于纤维品质形成的调控机制不清,造成种质资源利用靶向性不强,限制了优质棉育种的发展。因此,破解棉花种质资源多样性形成的亚基因组演化机制及调控网络重塑,解析At和Dt两个亚基因组如何协同调控纤维品质形成这一科学问题,将为纤维品质改良提供理论支撑。
棉花团队在过去的研究中发现了陆地棉亚基因组不对称驯化选择影响优质纤维形成(Nature Genetics, 2017),解析了棉花纤维“从无到有”演化和海岛棉特优纤维品质形成的遗传基础(Nature Genetics, 2019 & 2022),但对于两个亚基因组协同调控纤维品质形成的时空遗传机制认识十分有限。本研究通过对376份陆地棉的基因组和纤维发育6个时间点的2215份群体转录组数据进行联合分析,鉴定了引起不同材料间基因动态差异表达的遗传调控变异,包括53854个cis-eQTL与23811个trans-eQTL,分别调控18637与10391个基因,发掘了1258个与纤维品质性状(纤维长度、强度、整齐度、伸长率)相关的候选基因。候选基因和相关的遗传变异组成了36个调控模块,不同模块对于纤维品质性状形成的贡献存在差异,同一模块内基因在不同时期的表达量对性状的解释力也存在差异。
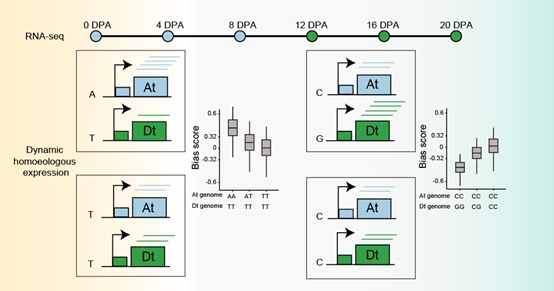
图1. 同源基因偏向性表达的eQTL分析模式图
棉花亚基因组同源基因在纤维发育不同时期会呈现亚基因组动态偏向性表达。为了解析亚基因组同源基因出现表达偏向的遗传机制,该研究创新地将同源基因的表达偏向程度量化为bias score,并以bias score作为表型与遗传变异进行关联分析,发掘与表达偏向相关的遗传变异(bias-eQTL;图1)。发现两个亚基因组同源基因同时被鉴定为纤维品质相关候选基因的比例极低(纤维长度0.8%,纤维强度2.7%,纤维整齐度0.8%,纤维伸长率3.7%),这意味着亚基因组同源基因对中未被鉴定为候选基因的一方(候选基因的同源基因)可能是潜在的育种改良基因资源。具有长纤维以及强纤维的材料倾向于积累更高比例的优异同源基因型(两个亚基因组同时被选择利用的同源基因对),而仅有~30%的同源基因对在每个时期50%以上的材料中被鉴定为优异同源基因型,从而解锁了同源基因在纤维改良中可以进一步被选择利用的巨大潜力。
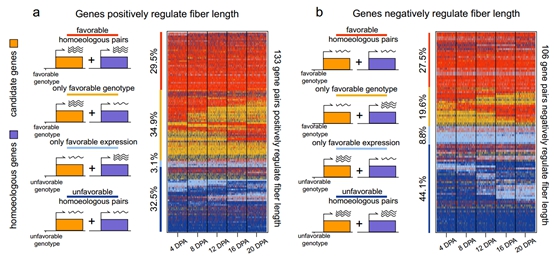
图2. 纤维长度相关同源基因在群体材料中的聚合利用情况
该研究利用纤维品质相关的调控变异和候选基因对3552份陆地棉种质资源进行育种评价,并训练模型开展纤维品质预测。研究发现,同时提供候选基因的基因型与同源基因的表达量可以显著提升纤维品质预测精度。基于此,评估了376份陆地棉材料两两杂交后有利变异的积累情况,并对杂交后代的纤维品质进行预测。例如,材料S003可以为S161提供一系列互补的有利变异,分布在8个调控模块,使两个亚基因组优异基因型得到有效互补聚合。预期用S003改良S161,可以使S161的纤维长度由28.77 mm提升至31.04 mm,纤维强度由29.72 cN/tex提升至37.66 cN/tex。这些结果为育种家精准选择亲本组合改良纤维品质提供了指导(图3)。在今后育种中,可以根据目标性状选择合适的模块,并通过对目标位点的聚合实现模块中更多优异基因的整合利用,即实现模块化的育种。
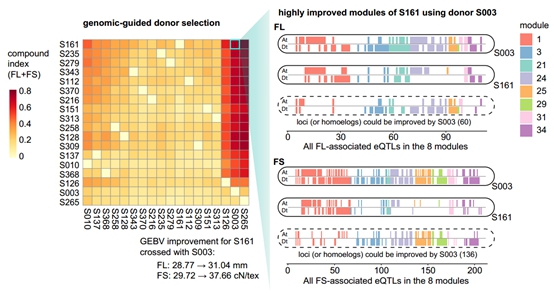
图3. 纤维品质改良的基因组模块化育种设计与亲本选配
总之,该研究阐明了亚基因组不对称选择的理论基础是来自于亚基因组优异基因型的不对称积累。其表现为性状相关调控位点仅有一个亚基因组出现优异基因型,或者尽管都存在但未同时被育种选择利用,从而导致亚基因组同源基因对性状形成的遗传贡献潜力未能最大化发掘。通过构建调控网络模块,训练模型对纤维品质进行预测,提出了亚基因组模块化的性状改良设计路线,为异源多倍体聚合重要调控模块开展性状设计育种提供了参考。
华中农业大学作物遗传改良全国重点实验室和湖北洪山实验室的王茂军、张献龙和涂礼莉教授为该论文的共同通讯作者,博士生尤佳琦、刘振平、戚正阳、博士后马益赞为该论文的共同第一作者。美国爱荷华州立大学Jonathan F. Wendel教授、美国农业部南方研究中心David D. Fang研究员和英国杜伦大学Keith Lindsey教授等团队或老师共同参与了研究。该研究中的田间实验在黄冈市农业科学院棉麦研究所李蔚团队的实验站完成。该研究得到了国家重点研发计划、国家自然科学基金、作物遗传改良全国重点实验室自主课题、湖北洪山实验室重大项目的资助。
英文摘要:
Polyploidy complicates transcriptional regulation and increases phenotypic diversity in organisms. The dynamics of genetic regulation of gene expression between coresident subgenomes in polyploids remains to be understood. Here we document the genetic regulation of fiber development in allotetraploid cotton Gossypium hirsutum by sequencing 376 genomes and 2,215 time-series transcriptomes. We characterize 1,258 genes comprising 36 genetic modules that control staged fiber development and uncover genetic components governing their partitioned expression relative to subgenomic duplicated genes (homoeologs). Only about 30% of fiber quality-related homoeologs show phenotypically favorable allele aggregation in cultivars, highlighting the potential for subgenome additivity in fiber improvement. We envision a genome-enabled breeding strategy, with particular attention to 48 favorable alleles related to fiber phenotypes that have been subjected to purifying selection during domestication. Our work delineates the dynamics of gene regulation during fiber development and highlights the potential of subgenomic coordination underpinning phenotypes in polyploid plants.
审核人 王茂军
论文链接:https://www.nature.com/articles/s41588-023-01530-8
延伸阅读:【新闻特写】在棉花亚基因组里“挖金矿”
