南湖新闻网讯(通讯员 王茂军) 近日,我校棉花遗传改良团队发表相关研究论文,首次公布了棉属中比四倍体棉花基因组更大的K2基因组,并对A2基因组和D5基因组进行了升级,发现基因组特异的转座子扩增导致了基因组扩张,通过比较三维基因组研究揭示了年轻的转座子扩增伴随着棉属特异的染色质高级结构形成。
棉花(Gossypium)家族成员众多,含有大约52个种,共9种基因组类型,包括二倍体基因组A-G、K和四倍体AD。二倍体棉种染色体数目一致,但基因组大小差异达三倍以上,最大的二倍体基因组K比四倍体基因组AD更大。因此,棉花是研究基因组大小进化的重要资源。该团队前期的研究表明,二倍体A和D基因组在多倍化形成四倍体AD基因组的过程中,伴随着染色质高级结构的重新组织,并对同源基因的表达调控产生影响,但对于转座子活动与染色质高级结构进化之间的关系知之甚少。
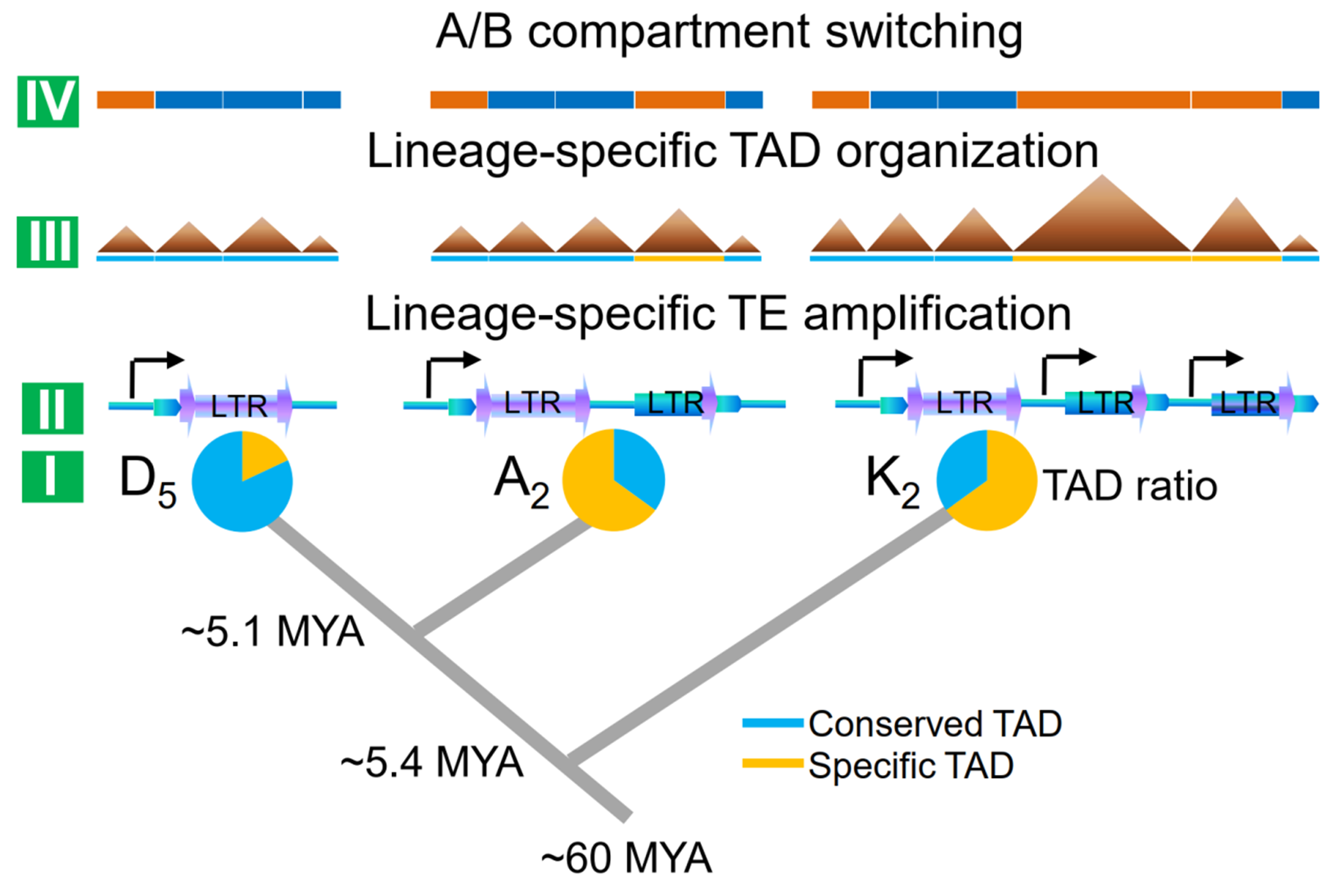
本研究利用Nanopore测序技术组装了圆叶棉的基因组(K2),组装大小为2444 Mb;提升了亚洲棉(A2)和雷蒙德氏棉(D5)的基因组,组装大小分别为1621 Mb和750 Mb。重复序列注释表明K2基因组中重复序列占81%,A2占68%,D5占57%。相对于D5,K2和A2中棉种特异的反转录转座子扩增是造成这三个基因组大小三倍变化的原因, 特别是Gypsy和DIRS类型,但转座子扩增对同源基因的位置关系影响较小。全长转座子插入时间分析表明K2基因组中转座子插入最为古老,A2基因组有更多年轻的转座子。
结合染色质互作数据,研究发现三个基因组中17%的共线性同源基因表现为A/B区室的染色质状态改变,这与活跃的转座子扩增相关。相对于D5基因组,在K2和A2基因组转座子的扩增伴随着A区室(活跃)的基因增多。同时,大约60%的拓扑结构域(TAD)在三个基因组中发生了重新组织,特别是K2基因组不保守的TAD边界存在特异的和年轻的转座子插入。基于这些结果,提出了三个棉种分化过程中,基因组扩张-转座子扩增介导的A/B 区室转换和TAD重组的进化模型(图1)。
棉花转座子扩增与染色质三维结构进化模型
该研究论文发表于国际学术期刊Molecular Biology and Evolution。我校王茂军教授和李健英博士为论文共同第一作者,张献龙教授、中国农业科学院棉花研究所王坤波研究员和爱荷华州立大学Jonathan F.Wendel教授为共同通讯作者。该研究得到了国家转基因专项和国家自然科学基金等项目的资助。
审核人:张献龙
【英文摘要】
Transposable element (TE) amplification has been recognized as a driving force mediating genome size expansion and evolution, but the consequences for shaping 3D genomic architecture remains largely unknown in plants. Here, we report reference-grade genome assemblies for three species of cotton ranging three-fold in genome size, namely Gossypium rotundifolium (K2), G. arboreum (A2), and G. raimondii (D5), using Oxford Nanopore Technologies. Comparative genome analyses document the details of lineage-specific TE amplification contributing to the large genome size differences (K2, 2.44 Gb; A2, 1.62 Gb; D5, 750.19 Mb), and indicate relatively conserved gene content and synteny relationships among genomes. We found that approximately 17% of syntenic genes exhibit chromatin status change between active (“A”) and inactive (“B”) compartments, and TE amplification was associated with the increase of the proportion of A compartment in gene regions (∼ 7,000 genes) in K2 and A2 relative to D5. Only 42% of topologically associating domain (TAD) boundaries were conserved among the three genomes. Our data implicate recent amplification of TEs following formation of lineage-specific TAD boundaries. This study sheds light on the role of transposon-mediated genome expansion in the evolution of higher-order chromatin structure in plants.
原文链接:

