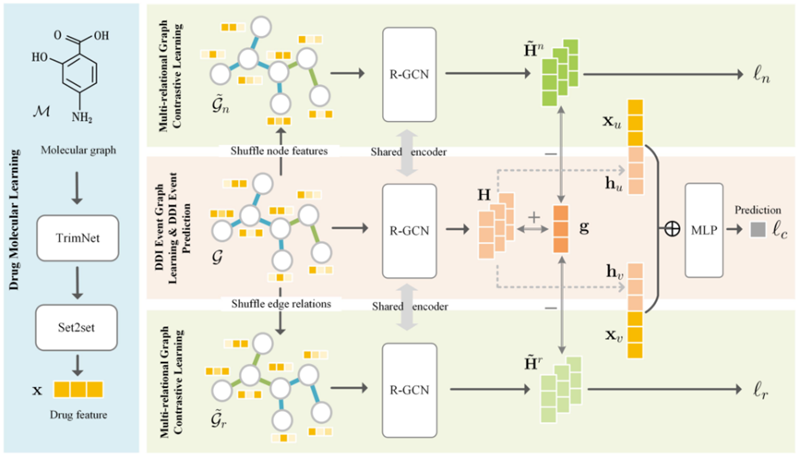
多关系对比学习图神经网络MRCGNN
南湖新闻网讯(通讯员 熊展坤)近日,我校信息学院人工智能与知识发现团队以“Multi-relational Contrastive Learning Graph Neural Network for Drug-drug Interaction Event Prediction”为题的论文被国际人工智能领域会议AAAI-2023(The 37th AAAI Conference on Artificial Intelligence)录用。研究团队提出了一种新的药物反应事件预测方法,解决了现有方法药物信息整合不全,罕见药物反应事件预测精度低的问题。
在同时使用多种药物来联合治疗复杂疾病时,药物间的相互作用可能会带来意想不到的不良后果,如药物疗效降低或药物毒性增加等,这些不良后果称为药物反应事件。如何精准预测药物反应事件,避免对病人造成伤害,产生巨额医疗费用,是近年来人工智能与药物发现领域的热点研究问题。当前药物反应事件预测方法通常单一地考虑药物分子结构信息或药物交互信息,且对于一些发生率较低的药物反应事件(称为罕见药物反应事件)的预测精度较低,这些都限制了药物反应事件预测模型的性能。
为解决上述问题,我校研究团队提出了一种名为“多关系对比学习图神经网络”的药物反应事件预测方法MRCGNN。该方法将药物分子结构信息和药物交互信息进行分层整合,并在药物反应事件关联图上使用新设计的,基于双视图负对应增强策略的多关系对比学习来捕获关于罕见药物反应事件的隐含信息。
研究团队将MRCGNN与仅使用药物分子结构信息和仅使用药物交互信息的基线方法进行了对比,预测准确度分别提升6.10%和3.99%。此外,MRCGNN在罕见事件预测任务中,较之前性能表现最好的对比方法,预测准确度提升了大约30.75%。实验表明,药物分子结构信息和药物交互信息的整合能够充分利用不同信息提升模型性能,而研究团队所设计的多关系对比学习框架能够有效增强对罕见药物反应事件的表征和预测能力。因此,MRCGNN方法相比于现有方法具有更高的药物反应事件预测精度,并且能够显著提高模型预测罕见药物反应事件的能力。
信息学院博士生熊展坤、刘世超老师和博士生黄锋为论文共同第一作者,信息学院章文教授为论文通讯作者。信息学院博士生王紫嫣、博士生刘旋和纽约州立大学Binghamton分校张仲非教授(IEEE Fellow)也参与了该研究工作。
审核人:章文
【英文摘要】
Drug-drug interactions (DDIs) could lead to various unexpected adverse consequences, so-called DDI events. Predicting DDI events can reduce the potential risk of combinatorial therapy and improve the safety of medication use, and has attracted much attention in the deep learning community. Recently, graph neural network (GNN)-based models have aroused broad interest and achieved satisfactory results in the DDI event prediction. Most existing GNN-based models ignore either drug structural information or drug interactive information, but both aspects of information are important for DDI event prediction. Furthermore, accurately predicting rare DDI events is hindered by their inadequate labeled instances. In this paper, we propose a new method, Multi-Relational Contrastive learning Graph Neural Network, MRCGNN for brevity, to predict DDI events. Specifically, MRCGNN integrates the two aspects of information by deploying a GNN on the multi-relational DDI event graph attributed with the drug features extracted from drug molecular graphs. Moreover, we implement a multi-relational graph contrastive learning with a designed dual-view negative counterpart augmentation strategy, to capture implicit information about rare DDI events. Extensive experiments on two datasets show that MRCGNN outperforms the state-of-the-art methods. Besides, we observe that MRCGNN achieves satisfactory performance when predicting rare DDI events.

