南湖新闻网讯(通讯员 杜泽臻)近日,华中农业大学果蔬园艺作物种质创新与利用全国重点实验室、湖北洪山实验室焦文标课题组在Genome Biology上发表了题为“A comprehensive benchmark of graph-based genetic variant genotyping algorithms on plant genomes for creating an accurate ensemble pipeline”的研究论文。
研究结果表明,基于图形泛基因组的遗传变异分型算法虽然可以提高变异分型的准确性,但由于植物基因组的复杂性,其在植物基因组变异分型上仍存在巨大挑战。该研究还开发了一套适用于植物的图形基因组变异分型软件EVG。该项泛基因组研究将为农艺作物种质资源基因型鉴定、作物精准分子育种等领域提供准确高效的算法工具。

高通量DNA测序技术的飞速发展,使得科研工作者能快速、大规模地构建植物基因组的遗传变异图谱,也促进了泛基因组、全基因组关联分析等研究领域的快速发展。这些研究通常需要对大量个体基因组的重测序数据进行变异分型。传统分型算法依赖短序列比对,即将个体基因组的短片段测序数据比对到单个线性参考基因组。然而,由于单个参考基因组的偏好性,部分来自基因组高度差异区域的测序读段往往不能正确比对。此外,传统方法难以利用短片段数据,对结构变异正确地进行分型。
有研究表明,基于图形泛基因组的分型算法,能整合多个基因组的序列信息,降低比对偏好性,提高比对准确度,进而提升变异分型的能力。目前,众多研究者已经开发了多种基于图形泛基因组理论的变异分型算法。这些算法主要基于两类策略,即利用读段比对或Kmer谱比较进行变异分型,包括vg、GraphTyper、BayesTyper、PanGenie等。然而,这些算法主要基于人类基因组开发,并未考虑到植物基因组自身的特点(如:重复序列高、杂合度高、存在多倍体等)。

图1 图形基因组算法在不同植物基因组上的整体性能
为了探究这些算法在植物基因组变异分型中的应用效果,研究者通过准备拟南芥、水稻、大豆、玉米、油菜(异源四倍体)和杏(杂合二倍体)的模拟数据和真实数据,系统性地测试了目前常用的8款图形基因组变异分型软件在不同基因组复杂度、测序读长与深度、基因组杂合度、变异断点误差等条件下的分型效果。结果表明,随着基因组大小和重复序列的增加,算法的分型召回率会出现不同程度的降低(图1);在读段长度为150bp、测序深度在10X以上时,多数软件能有较好的变异分型效果。另外,随着群体变异数量的增加,基因组图的索引将变得异常复杂,同时会消耗大量的计算资源。研究者还测试了不同杂合度的基因组(0%-2.35%),结果表明,基因组杂合度同样会影响到变异的分型。此外,部分软件在结构变异位点出现断点错误、较小变异(如SNP、indel)或重复序列时,对结构变异的分型能力会出现较明显的下降。
尽管现有的图形基因组分型算法并非在所有的测试条件下都表现出出色的基因分型性能,但它们在分型能力上仍有很高的互补性。为了整合不同算法的优势,研究者开发了一款适用于植物基因组的集成式分型软件EVG(Ensemble Variant Genotyper)。该软件可以根据测序深度、读长、基因组大小等信息,自动选择用于下游分型的图形基因组分型算法,并且对输入输出文件格式进行适当转换。对于每个变异位点的最终分型结果,EVG会根据变异的大小和位置进行聚类,并基于多个算法的分型结果来确定最终的基因型(图2)。最后,研究者使用上述同样的模拟数据和真实数据,对EVG的分型能力进行了全面测试。结果表明,在各项测试条件下,EVG的分型效果更为稳定,能同时保持较高的准确率和召回率,并且只需要10×的测序数据即可获得较好的分型效果。
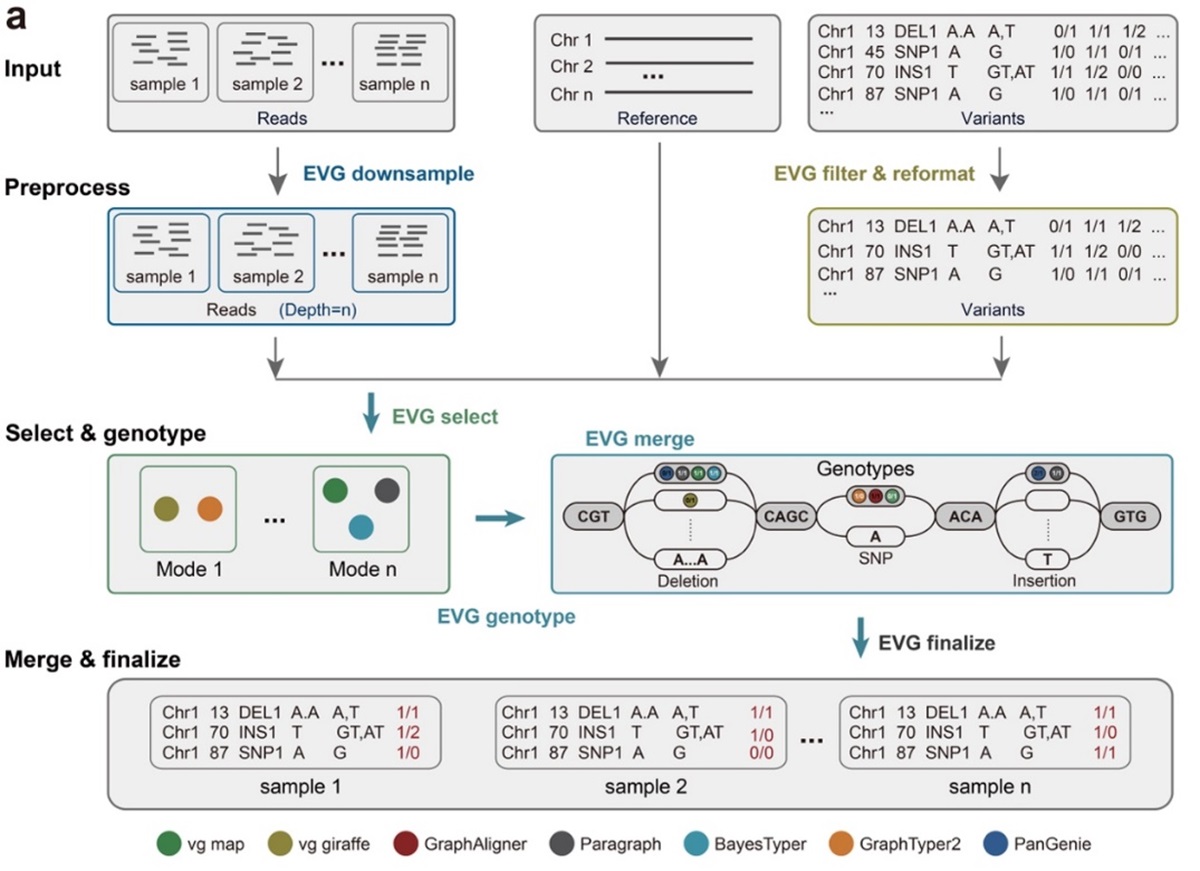
图2 EVG软件算法流程
综上所述,该研究揭示了现有图形基因组算法应用于植物基因组变异分型的重大挑战,包括资源消耗较高、重复序列相关变异分型较差和分型性能不稳定等。并且,研究者开发了适用于植物基因组的图形基因组集成式分型算法EVG。此算法有望用于构建基于低覆盖度重测序数据的作物大规模群体水平的遗传变异(包括结构变异等)图谱;推动植物基因泛基因组等领域的深入研究。
华中农业大学果蔬园艺作物种质创新与利用全国重点实验室、湖北洪山实验室、信息学院博士研究生杜泽臻为文章第一作者,焦文标教授为通讯作者。硕士研究生贺家宝也参与了该研究工作。该工作得到了国家自然科学基金、华中农业大学高层次人才启动基金的资助。
点击阅读原文
审核人:焦文标