南湖新闻网讯(通讯员 闫旭辉)北京时间2022年11月11日凌晨,湖北洪山实验室、作物遗传改良全国重点实验室、生命科学技术学院蛋白质科学研究团队联合北京大学唐淳教授课题组在《Cell Research》杂志在线发表题为“AI-empowered integrative structural characterization of m6A methyltransferase complex”的研究论文,报道了mRNA m6A甲基化转移酶复合体(m6A methyltransferase complex, 以下简称MTC)的分子全貌。
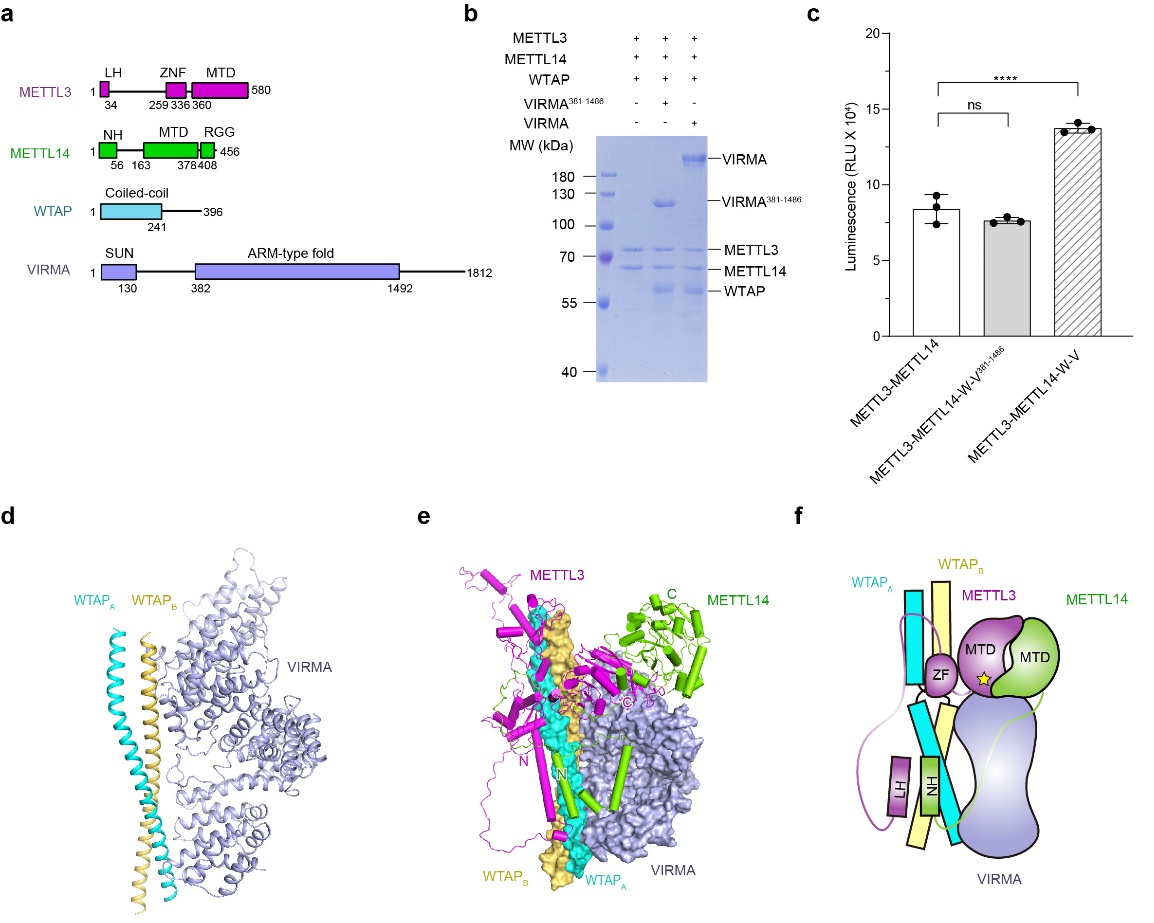
图1. m6A甲基化转移酶复合体(MTC)的分子全貌
RNA存在多种修饰,其中腺嘌呤N6位甲基化(m6A)修饰广泛存在于植物、动物和微生物中。m6A在转录后调控中发挥着关键作用,调控生物发育,与疾病发生相关。mRNA的m6A修饰由m6A甲基化转移酶复合体(MTC)催化产生,该复合体包含核心催化蛋白METTL3和METTL14,以及多个调节蛋白,包括WTAP、VIRMA、HAKAI、ZC3H13和RBM15等。尽管METTL3和METTL14催化机理的研究已经较为透彻,但是整体MTC运作机理并不清楚。完整MTC组成复杂且高度动态,是领域内研究其工作机理的主要难点。
研究人员探究了MTC复合体的组装形式,发现调控蛋白WTAP和VIRMA促进了甲基转移酶的催化活性,联合冷冻电镜、交联质谱和AI结构预测等多种研究手段,获得了核心MTC的分子全貌,展示了一种可能的MTC催化前的静息状态,提出了辅助蛋白对MTC的调控模型:WTAP作为“桥梁”连接VIRMA与METTL3-METTL14,VIRMA可为其他调节蛋白的结合提供“平台”,METTL3-METTL14催化“口袋”朝向VIRMA等待底物的到来(图1)。该模型提供了研究整体MTC催化过程的基础,以辅助蛋白为新靶点设计药物进行疾病治疗的新思路。
该研究团队于2016年在《Nature》报道了核心催化酶METTL3-METTL14催化结构域的运作基础,于2018年在《Protein & Cell》杂志报道了核心催化酶识别底物RNA的识别元件锌指结构域。此次在《Cell Research》报道的MTC分子全貌,是该研究团队聚焦RNA m6A甲基酶复合物作用机制研究的又一突破。
我校生科院博士研究生闫旭辉、官泽源博士和北京大学裴凯研究生为该论文共同第一作者,我校殷平教授和北京大学唐淳教授为共通讯作者。校级蛋白质平台和电镜平台为该研究的开展提供了强有力的支持。该研究受到了科技部项目、国家自然科学基金、湖北洪山实验室基金的资助。
原文链接:
https://www.nature.com/articles/s41422-022-00741-8
相关文章链接:
https://www.nature.com/articles/nature18298
https://link.springer.com/article/10.1007/s13238-018-0518-7
审核:殷平

