南湖新闻网讯(通讯员 于志超 周勇)3月21日和25日,作物遗传改良全国重点实验室、湖北洪山实验室张建伟教授课题组(https://zhang.hzau.edu.cn)联合阿卜杜拉国王科技大学(Rod A. Wing教授)、中国农业大学(郭伟龙副教授)、国际水稻研究所(Kenneth McNally研究员)、冷泉港实验室(Doreen Ware教授)以及亚利桑那大学,蒙彼利埃大学,聖安娜高等研究院等国内外高校与科研机构在《自然通讯》与《分子植物》连续发表两篇研究论文:“Pan-genome inversion index reveals evolutionary insights into the subpopulation structure of Asian rice”与“Rice Gene Index (RGI): a comprehensive pan-genome database for comparative and functional genomics of Asian rice”。这两项工作以代表亚洲稻群体结构的高质量基因组为研究对象,构建并全面分析基因组倒位图谱,建成首个基于同源基因簇的水稻泛基因组综合数据库。
亚洲栽培稻(Oryza sativa)是世界一半人口的主食。预计2060年至2070年世界人口将增加到约100亿,科学研究人员迫切需要培育产量高、营养丰富、绿色环保和气候适应性更强的新品种。据记载,全球共有多达五十万份水稻种质及其野生近缘种,而这些资源的基因组变异中(单核苷酸多态性、插入缺失、倒位和易位等)蕴藏着品种改良的重要宝藏,对其开展基于泛基因组的多组学研究具有重要意义。张建伟课题组致力于构建大量水稻铂金标准高质量参考基因组,在泛基因组水平挖掘大型结构变异在群体演化中的作用,搭建高效实用的泛基因组平台,以期为筛选水稻高产优质性状和精准育种奠定基础。
该课题组《自然通讯》论文详细报道了利用覆盖群体结构的73份亚洲稻(O. sativa, AA基因组)和2份野生稻(O. rufipogon 和O. Punctata)组成的高质量泛基因组,构建了泛基因组水平的稻属大片段基因组倒位结构变异图谱(Inversion Index)。该图谱含1769个非冗余倒位片段(>100bp),覆盖水稻日本晴29%的基因组序列。通过对比亚洲稻与野生稻发现其中有885个亚洲稻特有基因组倒位变异,96个O. rufipogon(AA基因组)特有倒位变异,322个O. punctata(BB基因组)特有倒位变异(图1)。演化史比较研究发现,基因组倒位发生频率可高达每百万年700次,远高于之前研究结果(图1)。此外,该研究还发现大量基因组倒位参与了调控水稻基因表达和遗传重组率等多项生命功能。
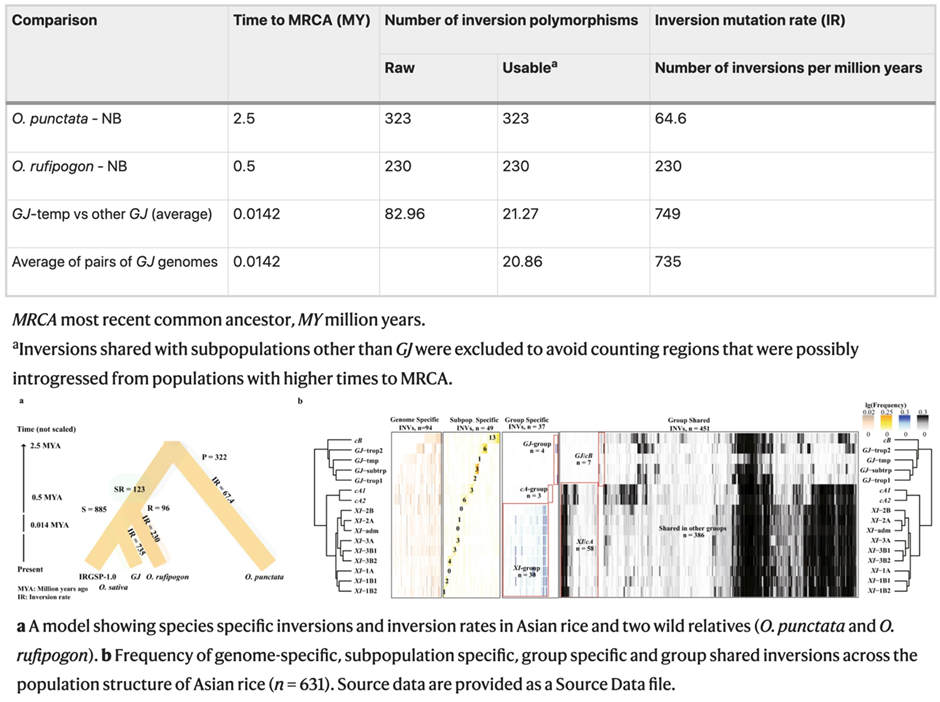
图1 泛基因组倒位变异频率评估和群体水平分布鉴定

图2 水稻基因索引数据库
同期,该课题组在《分子植物》介绍了通过整合代表性亚洲栽培稻种质基因组信息,进行综合比较分析建立首个基于同源基因簇的泛基因组数据库——水稻基因索引数据库(Rice Gene Index,RGI,图2A)。RGI为用户提供丰富的模块和工具,方便研究人员对不同种质基因以及基因同源关系进行查询、分析和可视化,为全球水稻研究学者提供免费在线检索和分析服务(图2B)。该平台为每个水稻基因建立一张“综合图文信息卡片(GeneCard)”实现不同功能和常用数据库之间的快速链接(图2C),记录包括OGI、基因通用名、序列、功能、表达等基本信息,并以可交互示意图展示该基因转录本结构、可变剪接事件、同源关系网络图和系统发育树等(图2D)。除此之外,研究人员可以通过关键词、序列(图2J)、同源关系(图2F)等三种方式在数据库中进行搜索,并在微观(图2E)和宏观(图2G)尺度对种质间基因组共线性进行分析和可视化。
上述两项工作是自2019年11月张建伟教授全职回国以来,依托华中农业大学水稻研究团队,立足生物信息技术平台,继2021年发布和解析全球首例水稻无缺口参考基因组后,对水稻泛基因组研究作出的最新成果,展现了我校在国际水稻研究中扮演的重要角色。为保证研究前沿性和准确性,张建伟教授多次带领课题成员与阿卜杜拉国王科技大学、亚利桑那大学、冷泉港实验室、中国农业大学等国内外单位同事合作交流。课题组凝练出“和趣求真 秉实生信”的发展理念,成员挑灯夜战,不懈探索,代表着我校师生和国内外同行“勤读力耕”的科研精神。这两项工作均受到华中农业大学启动费、自主科技创新基金项目和湖北洪山实验室等资助,并受华中农业大学作物遗传改良全国重点实验室生物信息计算平台的支持。
两篇文章相关信息如下:
《Pan-genome inversion index reveals evolutionary insights into the subpopulation structure of Asian rice》
阿卜杜拉国王科技大学博士后周勇、华中农业大学博士研究生于志超、国际水稻研究所研究员Dmytro Chebotarov和冷泉港实验室Kapeel Chougule为论文共同第一作者。阿卜杜拉国王科技大学Rod A. Wing教授、华中农业大学张建伟教授、国际水稻研究所Kenneth McNally研究员、冷泉港实验室Doreen Ware教授等为论文通讯作者。亚利桑那大学,蒙彼利埃大学,聖安娜高等研究院,Corteva Agriscience和Persephone参与该项研究。四川农业大学钦鹏教授对本研究提供了帮助和数据支持。
【英文摘要】
Understanding and exploiting genetic diversity is a key factor for the productive and stable production of rice. Here, we utilize 73 high-quality genomes that encompass the subpopulation structure of Asian rice (Oryza sativa), plus the genomes of two wild relatives (O. rufipogon and O. punctata), to build a pan-genome inversion index of 1769 non-redundant inversions that span an average of ~29% of the O. sativa cv. Nipponbare reference genome sequence. Using this index, we estimate an inversion rate of ~700 inversions per million years in Asian rice, which is 16 to 50 times higher than previously estimated for plants. Detailed analyses of these inversions show evidence of their effects on gene expression, recombination rate, and linkage disequilibrium. Our study uncovers the prevalence and scale of large inversions (≥100 bp) across the pan-genome of Asian rice and hints at their largely unexplored role in functional biology and crop performance.
原文链接:https://www.nature.com/articles/s41467-023-37004-y
《Rice Gene Index (RGI): a comprehensive pan-genome database for comparative and functional genomics of Asian rice》
华中农业大学博士研究生于志超、中国农业大学博士后陈永明和阿卜杜拉国王科技大学博士后周勇为论文共同第一作者。华中农业大学张建伟教授、中国农业大学郭伟龙副教授和阿卜杜拉国王科技大学Rod A. Wing教授为论文通讯作者。国际水稻研究所、冷泉港实验室、亚利桑那大学参与该项研究。
【英文摘要】
To integrate the genomic information of the rice pan-genome, we performed comparative analyses and established a user-friendly Rice Gene Index (RGI, https://riceome.hzau.edu.cn) platform with 16 platinum standard reference genomes and supplementary transcriptome data. To logically organize and scientifically the index of 744,233 genes among rice accessions, we detected 112,658 Ortholog Gene Indices, and provide ‘GeneCard’ pages to query genomic, transcriptomic, and homology information for each gene. The RGI allows users to search for relationships and comprehensive information of genes in keyword-based, sequence-based, and relationship-based ways. Furthermore, users can visualize these relationships at local and global scales corresponding to ‘Microcollinearity’ and ‘Macrocollinearity’ modules.
原文链接:https://www.cell.com/molecular-plant/fulltext/S1674-2052(23)00076-X
审核人 张建伟
