南湖新闻网讯(通讯员 王席 )玉米重要农艺性状的遗传解析对作物性状的遗传改良和全球的粮食安全非常重要。玉米雄穗分枝数是现代玉米育种过程中重要的选择目标,由众多微效的数量性状位点(QTL)控制。到目前为止,仍然缺乏快速批量克隆数量性状位点的方法,这严重限制了玉米雄穗分枝数性状的系统解析,一定程度上阻碍了现代玉米分子育种的发展。
8月26日,华中农业大学植物科学技术学院李林教授课题组在Nature Communications在线发表了题为“QTG-Miner aids rapid dissection of the genetic base of tassel branch number in maize” 的研究论文,开发了一种快速批量克隆作物数量性状位点的新方法QTG-Miner,定位并克隆了7个玉米雄穗分枝数功能基因,构建了玉米雄穗分枝数的分子调控网络,并揭示了现代玉米遗传改良过程中雄穗分枝数性状的驯化选择情况。
QTG-Miner方法的流程包括QTL的初定位、单个QTL位点分离材料的筛选和测序及利用QTG-Miner进行候选基因的挖掘三个步骤(图1)。
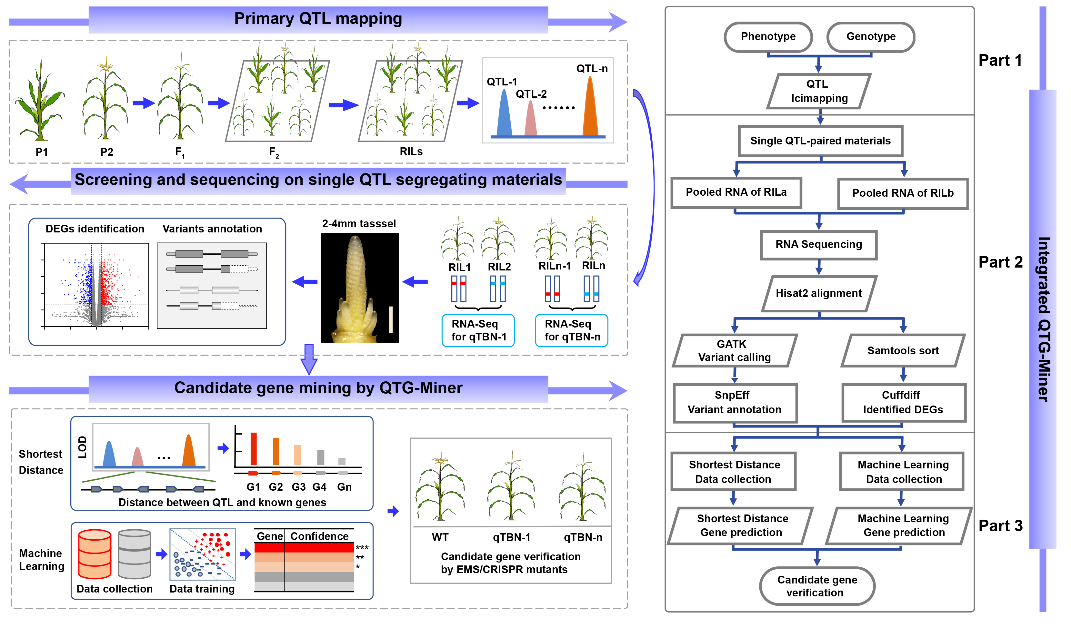
图1 QTG-Miner的流程
利用QTG-Miner方法,作者实现了快速批量克隆玉米雄穗分枝数QTL位点,克隆了7个雄穗分枝数QTL位点的功能基因。利用EMS突变体对上述基因进行了表型验证。同时作者也对其中的两个基因(ZmKinesin和Zmhd-zip120)进行了基因敲除实验,进一步证实了候选基因的真实性(图2)。
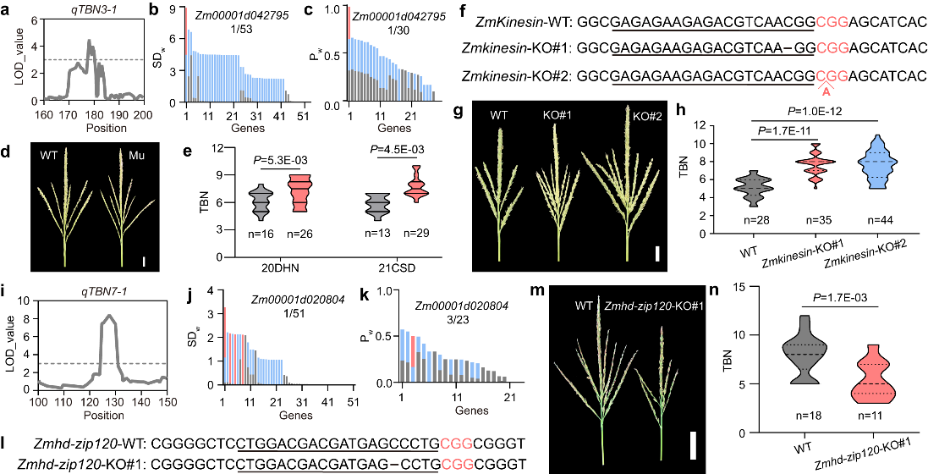
图2 qTBN3-1和qTBN7-1候选基因的验证
进一步,作者构建了玉米雄穗分枝数的分子调控网络,GO富集分析发现网络中的基因显著富集在多个生物学途径中。新的雄穗分枝数分子调控网络为从全局层面理解雄穗分枝数性状的发育机理提供了思路,也为新基因的挖掘提供了方向(图3)。
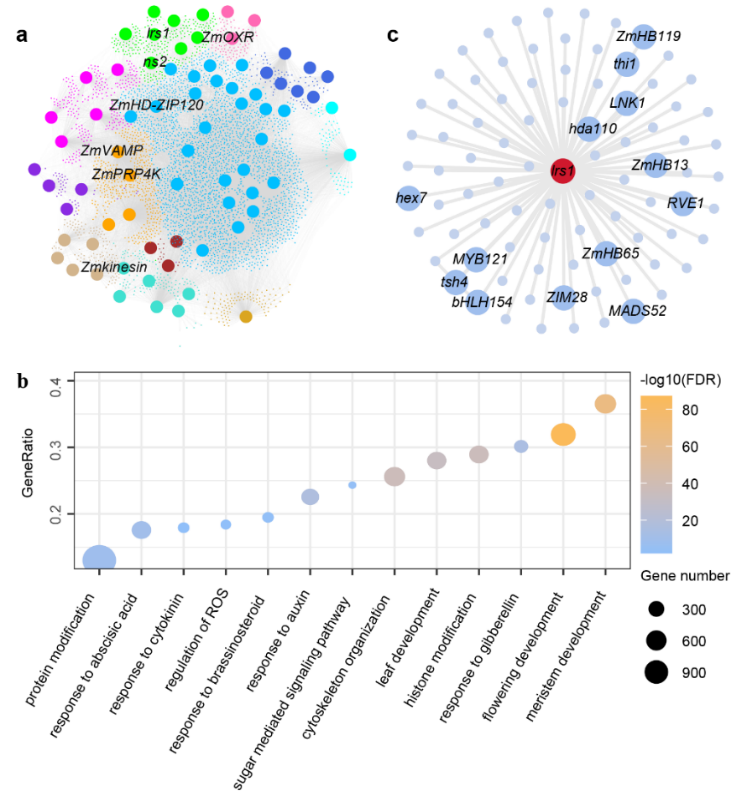
图3 玉米雄穗分枝数分子网络图谱
作者也探究了现代玉米育种过程中雄穗分枝数基因的选择历程。以lrs1基因为例,该基因在现代玉米改良的过程中受到了强烈的选择,序列多态性显著降低,优良等位基因型在玉米中的占比显著增加,也使得现代育种自交系的雄穗分枝数持续减少。
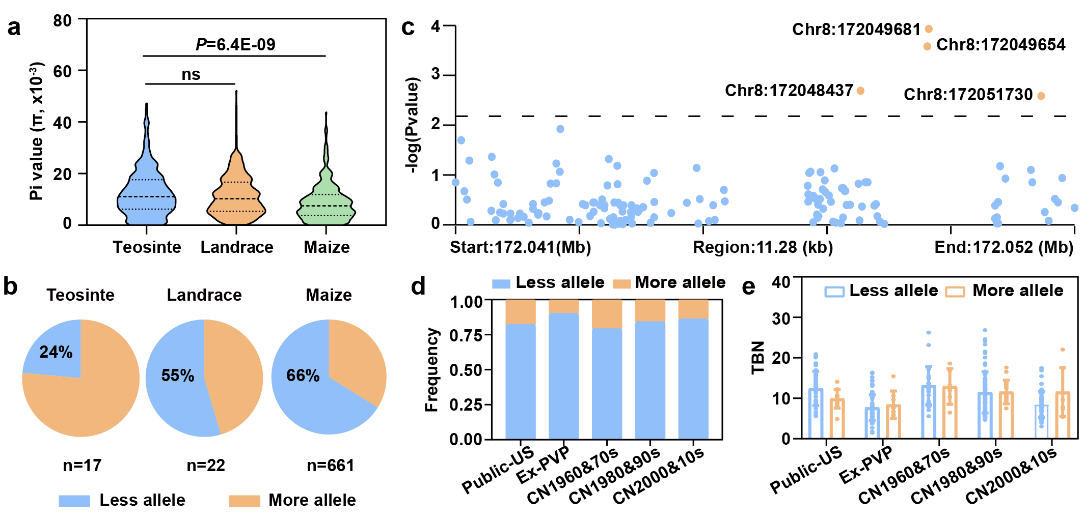
图4 lrs1基因的选择分析
该研究开发了一种快速批量克隆作物数量性状位点的新方法QTG-Miner,为系统解析作物重要农艺性状的遗传基础提供了方法和思路。该方法同样适用于玉米以及其他物种的多个农艺性状,具有非常广泛的应用前景。华中农业大学博士研究生王席与李娟为论文的共同第一作者,华中农业大学李林教授为通讯作者。感谢齐鲁师范学院路小铎教授和中国农科院李慧慧研究员对于本课题提供的帮助和建议,感谢仲恺农业工程学院齐永文教授在原始数据收集上的帮助,感谢中国农科院张春义研究员和齐鲁师范学院路小铎教授构建的玉米EMS诱变突变体库。该研究得到了国家自然科学基金项目、海南省崖州湾种子实验室项目、湖北洪山实验室重大项目、华中农业大学科技自主创新基金项目的资助。
李林课题组聚焦于玉蜀黍系统生物学研究,利用生物大数据与人工智能算法,系统解析了玉米密植高产的株型遗传基础,开发了作物精准智能设计育种体系CropGPT,服务于我国农作物生物种业。自2016年回国组建独立课题组以来,以通讯作者在Nature Genetics, Nature Biotechnology, Genome Biology, Molecular Plant (4篇),Nature Communications等国际主流期刊上发表论文近30篇。目前,正在从事玉米数字植物、作物泛功能基因组、超大群体杂种优势机制解析与精准设计育种、多年生分子机制解析与分子育种等非常有前景的研究项目,欢迎广大博士应聘该课题组博士后职位。
【英文摘要】
Genetic dissection of agronomic traits is important for crop improvement and global food security. Phenotypic variation of tassel branch number (TBN), a major breeding target, is controlled by many quantitative trait loci (QTLs). The lack of large-scale QTL cloning methodology constrains the systematic dissection of TBN, which hinders modern maize breeding. Here, we devise QTG-Miner, a multi-omics data-based technique for large-scale and rapid cloning of quantitative trait genes (QTGs) in maize. Using QTG-Miner, we clone and verify seven genes underlying seven TBN QTLs. Compared to conventional methods, QTG-Miner performs well for both major- and minor-effect TBN QTLs. Selection analysis indicates that a substantial number of genes and network modules have been subjected to selection during maize improvement. Selection signatures are significantly enriched in multiple biological pathways between female heterotic groups and male heterotic groups. In summary, QTG-Miner provides a large-scale approach for rapid cloning of QTGs in crops and dissects the genetic base of TBN for further maize breeding.
论文链接:https://www.nature.com/articles/s41467-023-41022-1
审核:李林
