南湖新闻网讯(通讯员 龙跃轩)近日,华中农业大学棉花团队受邀在植物学杂志Trends in Plant Science上发表综述论文“Evolutionary insights into the organization of chromatin structure and landscape of transcriptional regulation in plants”。
文章总结了植物中调控演化的研究进展,重点关注了植物调控元件的演化,并介绍了转座子驱动的顺式调控元件(CRE)演化模型。同时,研究人员讨论了染色质高级结构的演化模式,包括全基因组相互作用、不同层级的染色质结构以及同源基因的空间位置关系。此外,研究人员提出了未来调控演化的研究方向和需解决的重要问题。
研究人员着重阐述了植物多倍化过程对CRE的塑造模型。以小麦为例,在二倍体祖先A、B和D基因组分化期间,TE家族的差异化扩增塑造了CRE的多样性,导致多倍体小麦中转录因子的不平衡活动(图1B)。相对应,有一些在TE内部的CRE在二倍体分化和多倍化过程中都是保守的,在纯化选择下,即使这些CRE周围的序列已经发生较大变异(TE结构受到破坏),核心CRE也被保留了下来。
这些CRE的多样性在物种的形成和适应性演化上同样也具有十分重要的意义。在油菜中,CRE序列的多态性可能和开花关键基因BnaTFL1的四个拷贝完全不同的表达模式相关。淹涝相关的CRE基序增多和开放程度的增加有利于水生植物向陆生植物的演变。光合作用相关的重要转录因子GLK在不同的植物之间的结合位点也具有较大的特异性。
远端的CRE往往通过形成“染色质环”结构来调控基因的表达,因此研究基因的染色质高级结构对于我们理解调控演化具有重要的作用。在植物中,染色质的层级结构主要分为染色质疆域、A/B区室、拓扑关联结构域(TAD)和染色质环(loop)。
在全基因组互作层面,对于染色体内部互作来说,存在祖先染色体互作保留的现象(图1C)。来自同一个祖先的染色体片段间的互作要大于来自不同祖先染色体的片段间的互作。但随着物种形成后的演化,也会出现许多新的染色体互作。但对于不同的染色体间互作,来自相同和不同祖先的染色体互作没有显著的差异。在A/B区室层面,这一结构在演化中较为保守,这和祖先染色体保留现象相类似。但仍观察到大量的A/B区室转换事件,例如在Brassica rapa和Brassica oleracea之间,观察到了约有60%的区室转化事件。这些区室转换事件可能和基因组结构变异相关。
TAD在协调基因的表达方面起到了重要的作用。在演化中,TAD结构呈现较大的动态性。染色体重排富集在TAD边界内,然而SNV、InDel和PAV在TAD边界上减少。发生在TAD边界的转座子扩增和结构变异可能会介导TAD结构变化(新的TAD形成和TAD的融合);发生在TAD内部的不平衡的结构变异会导致TAD大小的变化(图1D)。植物中关于loop的演化研究较少。目前的研究表明物种特异的loop产生主要有三种因素(图1E)。第一,特异的序列导致特异的loop锚点;第二,loop锚点的染色质状态的改变;第三:序列变异介导的loop锚点间距离的变化。除此之外,在多倍化后,同源基因之间在空间位置上相对于其他基因之间较为接近,并且表达量较高的同源拷贝具有更多的染色质互作(图1F)。
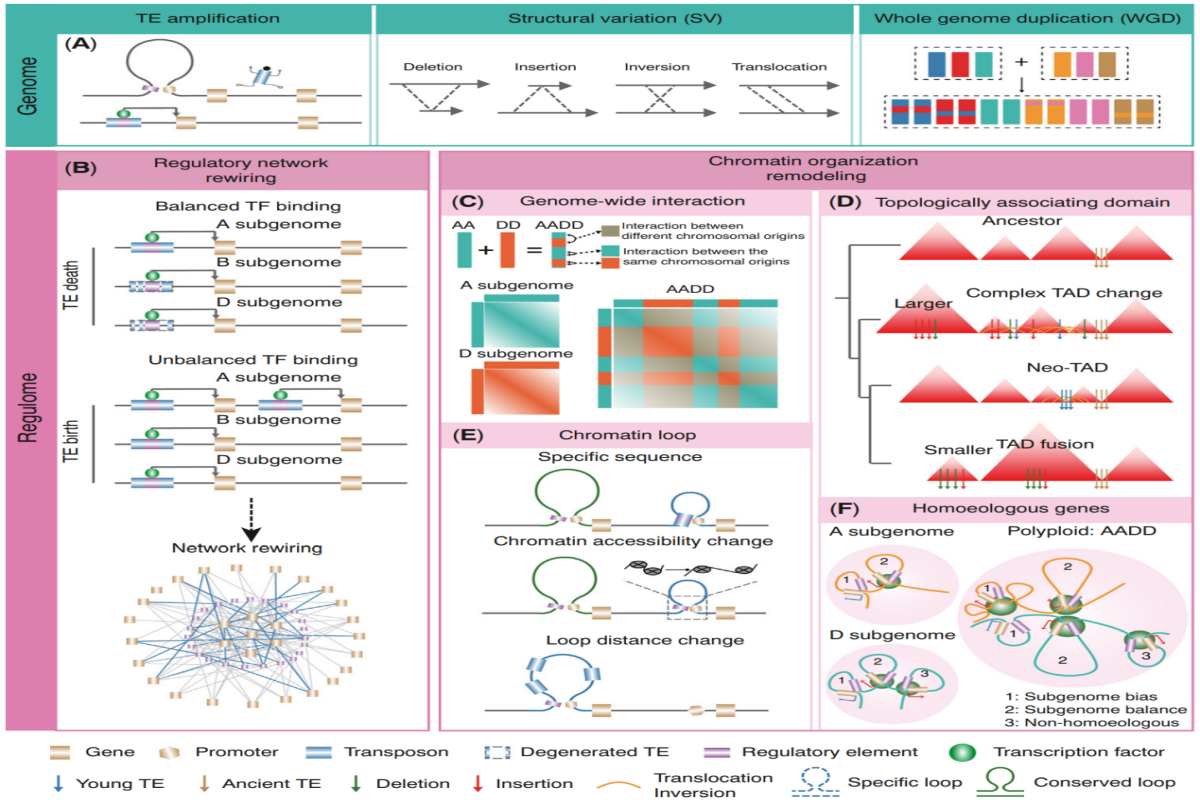
图1 序列水平变异关联的染色质高级结构和调控网络的重排
最后,研究人员对于未来调控演化的研究展开了四个方面的展望并提出了六个需要进一步解决的重要问题。
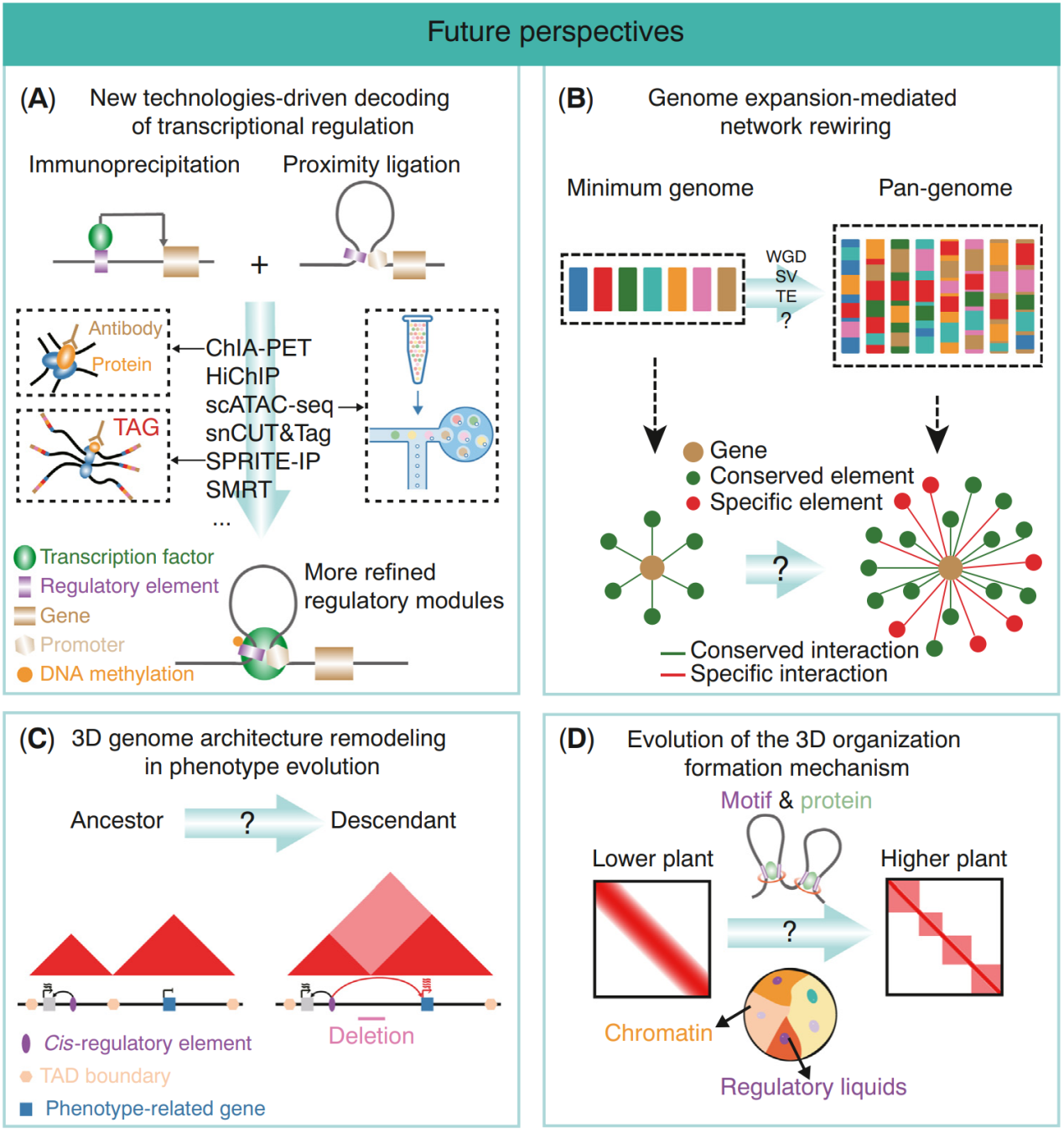
图2 调控元件和染色质高级结构演化的未来研究方向
华中农业大学硕士生龙跃轩为该论文的第一作者,王茂军教授为该论文的通讯作者。华中农业大学张献龙院士和美国爱荷华州立大学Jonathan F. Wendel院士参与了文章撰写和讨论工作。复旦大学张一婧教授和密歇根州立大学蒋继明教授对本文撰写提供了帮助。该工作得到了国家自然科学基金、国家重点研发计划、美国国家科学基金会植物基因组计划等项目的资助。
审核人 王茂军
Abstract
Development of complex traits necessitates the functioning and coordination of intricate regulatory networks involving multiple genes. Understanding 3D chromatin structure can facilitate insight into the regulation of gene expression by regulatory elements. This potential, of visualizing the role of chromatin organization in the evolution and function of regulatory elements, remains largely unexplored. Here, we describe new perspectives that arise from the dual considerations of sequence variation of regulatory elements and chromatin structure, with a special focus on whole-genome doubling or polyploidy. We underscore the significance of hierarchical chromatin organization in gene regulation during evolution. In addition, we describe strategies for exploring chromatin organization in future investigations of regulatory evolution in plants, enabling insights into the evolutionary influence of regulatory elements on gene expression and, hence, phenotypes.
