南湖新闻网讯(通讯员 赵虎)5月25日,我校水稻团队谢为博教授课题组发布了一项基于深度学习模型预测植物基因组序列变异调控效应的网络服务,该服务还可用于分析序列中的顺式调控元件。相关研究成果发表在国际学术期刊Nucleic Acids Research上。
全基因组关联分析(GWAS)被广泛用于农艺性状重要调控位点的鉴定,然而GWAS获得的显著位点并不一定是关键变异位点。此外,如何将GWAS结果与调控机制联系起来,目前仍充满挑战。通过变异(尤其是非编码调控区域的变异)效应的预测,可帮助筛选GWAS结果中的关键变异以及候选基因的鉴定。
真核生物中核DNA被紧密的包装成核小体,转录因子与DNA互作和行使转录调控功能将导致核小体解离,形成染色质开放区(open chromatin region,OCR)。因此,染色质开放状态是影响基因表达的重要表观遗传因素。近年来,多项人类医学中的研究表明,深度神经网络模型可从基因组序列中提取复杂特征,这些特征部分代表了顺式调控元件,因而可准确预测染色质开放状态。利用染色质开放状态数据训练获得的深度神经网络模型,可评估非编码区序列变异的效应及鉴定可能的顺式调控元件。其原理是,利用模型分别预测非编码序列变异对应的两个单倍型染色质开放状态,如两个状态间差异较大,表明该变异可能具有较大效应。当对一段非编码序列中的每个碱基进行饱和突变(in silico saturated mutagenesis)并预测其效应,可鉴定出一些连续高影响力位点,有研究表明这些位点往往代表了顺式调控元件。
尽管此类方法在人类医学中已得到一定应用,但这一领域的研究在植物中仍关注较少。此外,深度学习模型的构建需花费大量计算资源,很难被普通实验研究者直接利用。因此,植物中亟需可以帮助研究者快速评估序列变异调控效应以及鉴定序列中的调控元件在线网络服务。
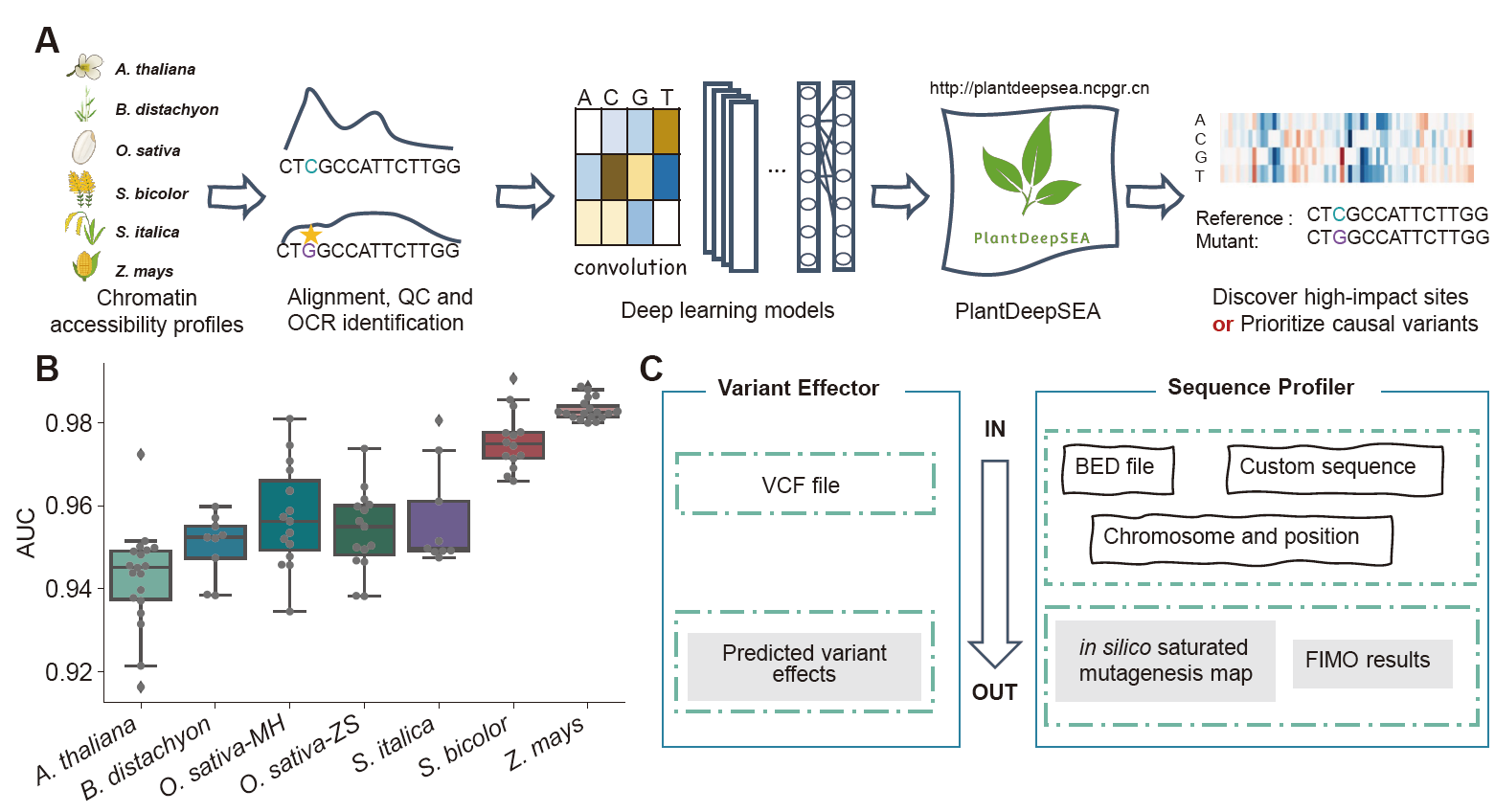
该研究中,谢为博教授课题组通过ATAC-seq技术获得5种禾本科植物物种(包含4种作物)多个组织的高质量染色质可及性数据,并收集了已发表拟南芥数据,从中鉴定了OCR。利用各个物种对应的参考基因组序列以及OCR信息,使用深度学习模型框架DeepSEA进行训练。通过评估,各模型曲线下面积(AUROC)在0.93-0.99之间,表明所构建的系列模型具有较高可用性。
谢为博教授课题组基于所获得的深度学习模型,构建了PlantDeepSEA(http://plantdeepsea.ncpgr.cn)网络服务,免费供所有用户使用。该网站主要包含2项功能:一是Variant Effector,其目的是预测序列变异对于各组织中染色质可及性的影响,用户可提交VCF文件进行变异效应预测;一是Sequence Profiler,该工具利用"in silico saturated mutagenesis"挖掘序列中的高影响力位点(High-impact sites,如顺式调控元件)。用户可选择染色体编号和输入基因组坐标、任意一段核苷酸序列或者包含一个或多个染色体区段的BED文件进行分析。由于计算资源所限,目前PlantDeepSEA允许1次提交最多2000个变异位点进行变异效应预测,以及5个区段进行调控元件分析。
我校博士后赵虎、硕士研究生涂卓为该论文共同第一作者,信息学院谢为博教授为该论文通讯作者,胡学海教授指导了部分研究工作。该研究数据分析工作得到武汉大学超算平台及华中农大作物遗传改良国家重点实验室生物信息计算平台支持和帮助,得到国家重点研发计划(2016YFD0100803)与国家自然科学基金(31771755, 31922065)资助。
英文摘要
Characterizing regulatory effects of genomic variants in plants remains a challenge. Although several tools based on deep-learning models and large-scale chromatin-profiling data have been available to predict regulatory elements and variant effects, no dedicated tools or web services have been reported in plants. Here, we present PlantDeepSEA as a deep learning-based web service to predict regulatory effects of genomic variants in multiple tissues of six plant species (including four crops). PlantDeepSEA provides two main functions. One is called Variant Effector, which aims to predict the effects of sequence variants on chromatin accessibility. Another is Sequence Profiler, a utility that performs ‘in silico saturated mutagenesis’ analysis to discover high-impact sites (e.g., cis-regulatory elements) within a sequence. When validated on independent test sets, the area under receiver operating characteristic curve of deep learning models in PlantDeepSEA ranges from 0.93 to 0.99. We demonstrate the usability of the web service with two examples. PlantDeepSEA could help to prioritize regulatory causal variants and might improve our understanding of their mechanisms of action in different tissues in plants. PlantDeepSEA is available at http://plantdeepsea.ncpgr.cn/.
论文链接:
https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkab383/6284177
审核人:谢为博

