
Figure 1 甜橙芽变(体细胞变异)群体的表型
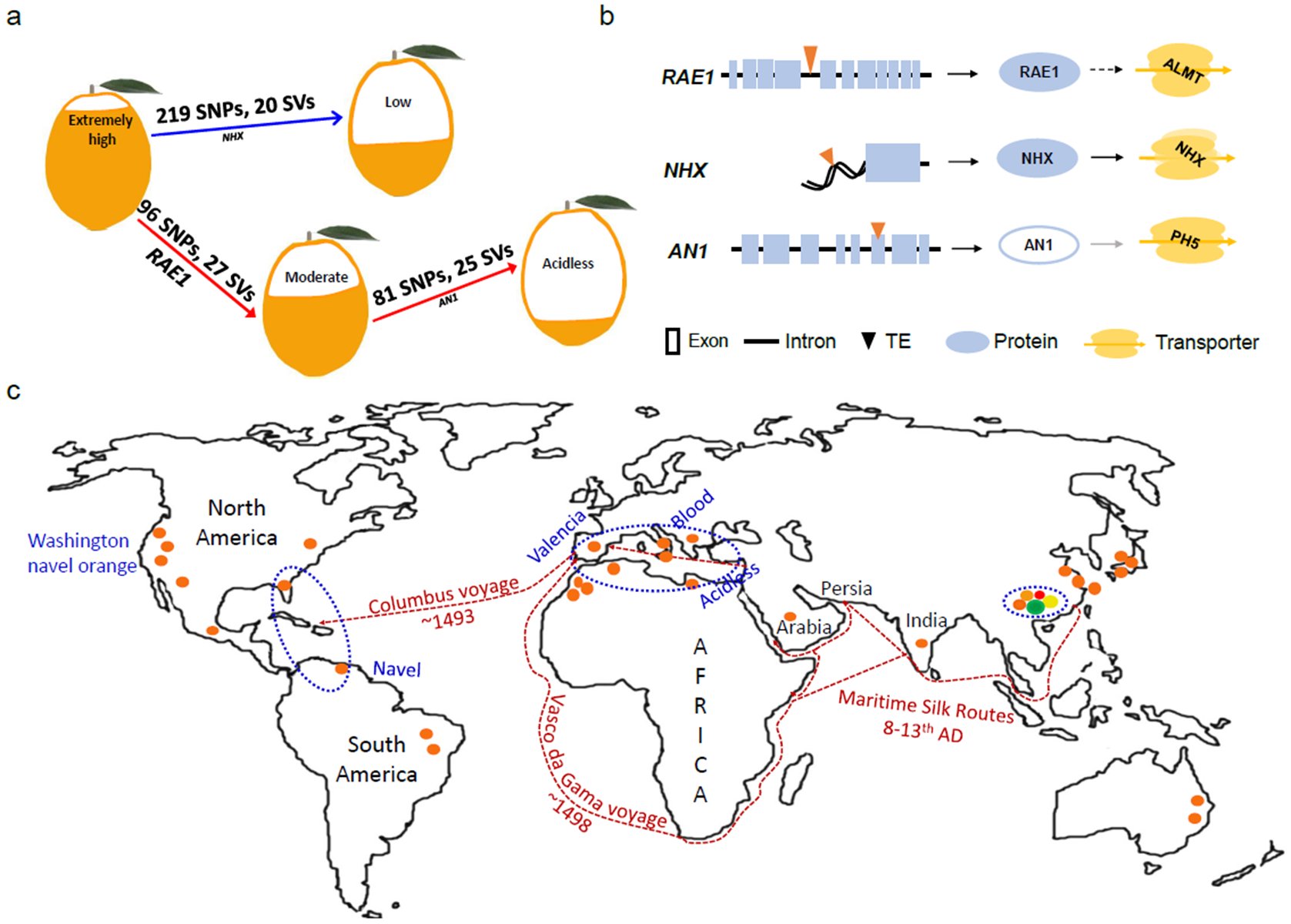
Figure 2 甜橙群体果实柠檬酸含量的分化(a)、其遗传基础(b)以及可能的传播路线(c)
南湖新闻网讯(通讯员 王沦)6月17日,我校园艺林学学院徐强课题组发现了影响甜橙果实酸味的体细胞变异。相关研究论文以题为“Somatic variations led to the selection of acidic and acidless orange cultivars”发表在Nature Plants。
大多数果树通过无性繁殖,基于体细胞变异的芽变育种成为果树重要育种途径。据统计,柑橘家族60%的品种来源于芽变育种。甜橙在全世界114个国家栽培,绝大多数品种来源于体细胞变异。甜橙具有无融合生殖特点,自然条件下进行严格的无性繁殖,成为体细胞变异研究的模式物种。体细胞变异来源于细胞有丝分裂过程中DNA复制和修复产生的一定概率的突变。相比于有性变异,人们对植物体细胞变异认识较少。在人类疾病中,体细胞变异的累积与癌症发生密切相关。在果树育种中,体细胞变异被广泛利用于芽变选种。
柠檬酸是柑橘果实风味的关键因子,团队前期研究发现糖酸比是柑橘果实风味的决定因子,柑橘驯化过程中主要降低了柠檬酸含量而改良了口感。该研究发现甜橙114个芽变材料的果实柠檬酸含量变化十分显著,如我国湖南南部收集到的自然高酸甜橙材料,其果实酸的含量接近于柠檬;而无酸甜橙的柠檬酸含量接近为0。
为了解析甜橙体细胞变异的遗传基础,挖掘影响果实酸味的关键基因,该研究采用了芽变群体结合基因组学的策略,提高了体细胞预测范围和精准度。研究团队首先组装了甜橙双单倍体(Double Haploid)及6个二倍体的基因组,构建起迄今柑橘家族质量最好的参考基因组,9条染色体平均每条染色体仅剩下3个缺口(gap)。利用高质量基因组和丰富的芽变材料,该研究在114个甜橙中共鉴定到 2321个结构变异(Structure variation),进一步鉴定到877个转座子跳跃事件。遗传和基因功能研究表明,这些转座子在低酸和无酸的突变体中插入到转运子(Transporter)基因或者其调控基因,从而影响了果实的pH值及柠檬酸含量。
甜橙在历史上数次从中国经过海上丝绸之路、陆地茶马古道传播到外地,据报道,最初被波斯(今伊朗地区)、地中海地区商人引入的中国甜橙是高酸类型,这类高酸的果实易于长途储运但风味不好。后来从中国引进了酸含量中等、风味更好的甜橙类型,并命名为China orange。本研究的遗传变异也支撑甜橙在历史上的传播路线,并在地中海品种和美洲品种检测到了更多的体细胞变异。本研究结果首次阐明了甜橙体细胞变异的基因组基础,为芽变机制提供了理论支撑,同时也为果实风味育种提供了基因资源。
我校园艺林学学院博士后王沦、博士生黄跃和刘子昂为该论文的共同第一作者,徐强教授为论文的通讯作者,邓秀新院士参与了项目的指导。西南大学柑橘研究所江东研究员、湖南农科院园艺所杨水芝研究员、陈鹏助理研究员参与了研究。该研究获得国家重点研发计划,国家自然科学基金,中央高校基础科研基金等基金的资助。
Abstract
Somatic variations are a major source of genetic diversification in asexual plants, and underpin clonal evolution and the breeding of asexual crops. Sweet orange is a model species for studying somatic variation because it reproduces asexually through apomixis and is propagated asexually through grafting. To dissect the genomic basis of somatic variation, we de novo assembled a reference genome of sweet orange with an average of three gaps per chromosome and a N50 contig of 24.2 Mb, as well as six diploid genomes of somatic mutants of sweet oranges. We then sequenced 114 somatic mutants with an average genome coverage of 41×. Categorization of the somatic variations yielded insights into the single-nucleotide somatic mutations, structural variations and transposable element (TE) transpositions. We detected 877 TE insertions, and found TE insertions in the transporter or its regulatory genes associated with variation in fruit acidity. Comparative genomic analysis of sweet oranges from three diversity centres supported a dispersal from South China to the Mediterranean region and to the Americas. This study provides a global view on the somatic variations, the diversification and dispersal history of sweet orange and a set of candidate genes that will be useful for improving fruit taste and flavour.
原文链接:https://www.nature.com/articles/s41477-021-00941-x
审核人 徐强

